上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext单细胞/超低起始量 cDNA 合成 &扩增模块 收藏
Download:
- isoschizomers |
- compatible ends |
- single letter code
特性
概述
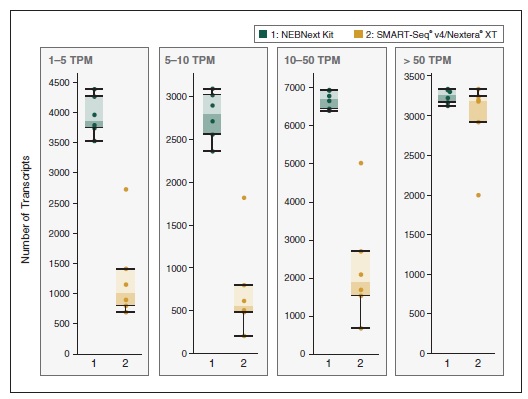
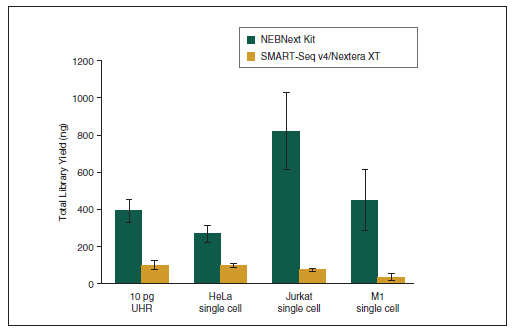

上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。


上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 多样本接头引物试剂盒 2(96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 3 (96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 4 (96 种 Unique 双端)
-20°C
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 多样本接头引物试剂盒 1(96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 3 (96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 4 (96 种 Unique 双端)
-20°C
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 多样本接头引物试剂盒 1(96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 2(96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 4 (96 种 Unique 双端)
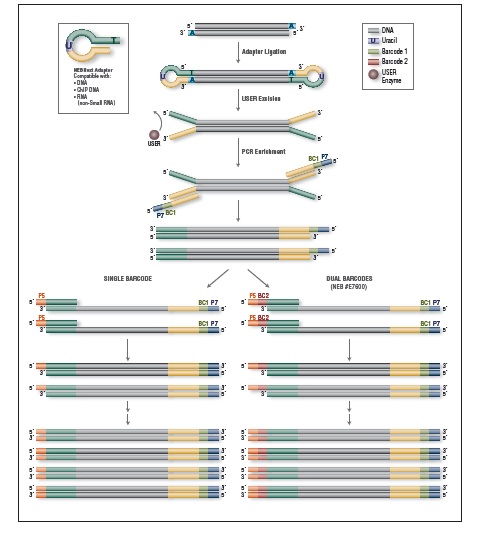
-20°C
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 多样本接头引物试剂盒 1(96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 2(96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 3 (96 种 Unique 双端)
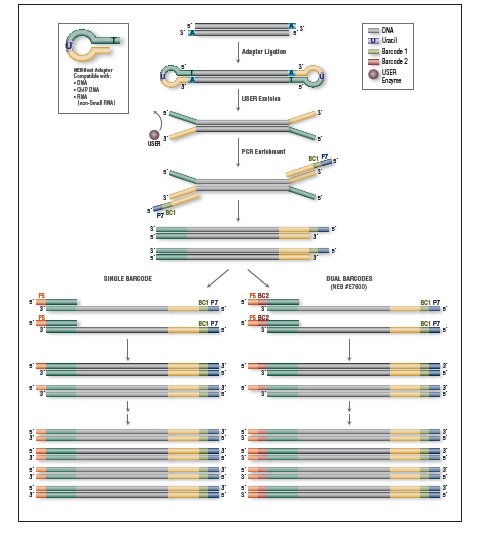
-20°C
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 多样本接头引物试剂盒 1(96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 2(96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 3 (96 种 Unique 双端)
NEBNext 多样本接头引物试剂盒 4 (96 种 Unique 双端)
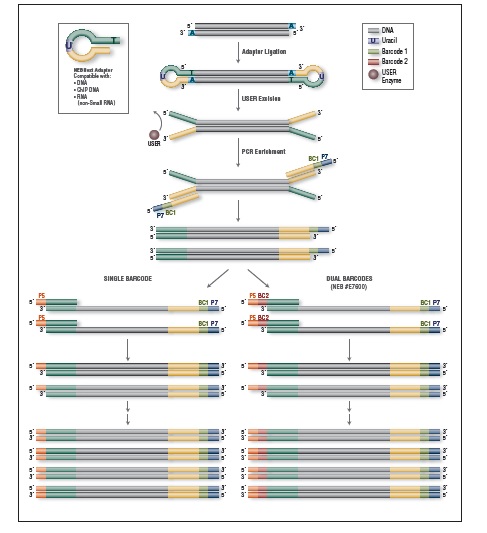
-20°C
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
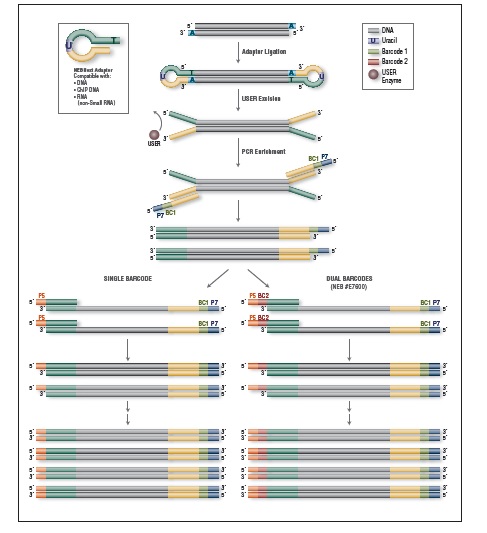
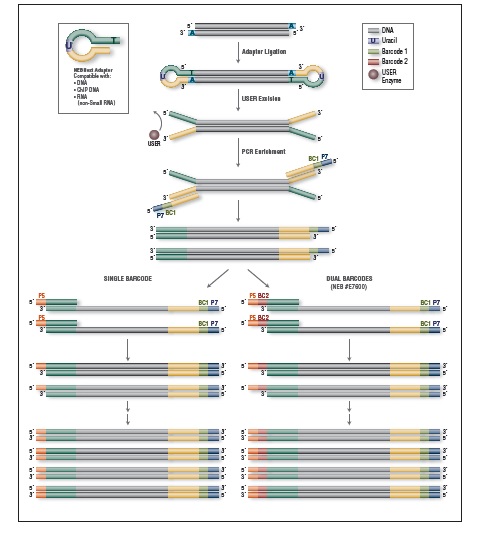
·提高连接效率
·极大降低接头二聚体
·增加文库产量
·增加样品识别特异性 (双端检索)
·大量单端检索/可用检索
·提供检索表和样本示例表
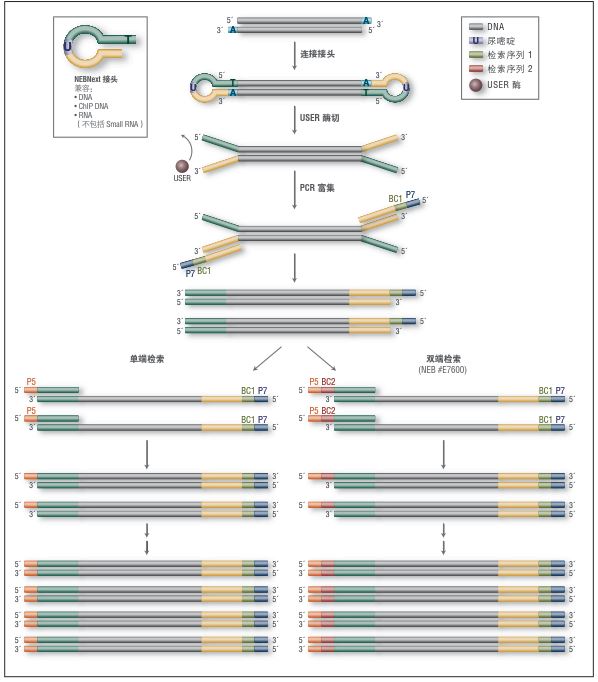
每个试剂盒都通过对基因组 DNA、ChIP DNA 和 mRNA 进行文库构建,并在 Illumina 平台测序进行验证。
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
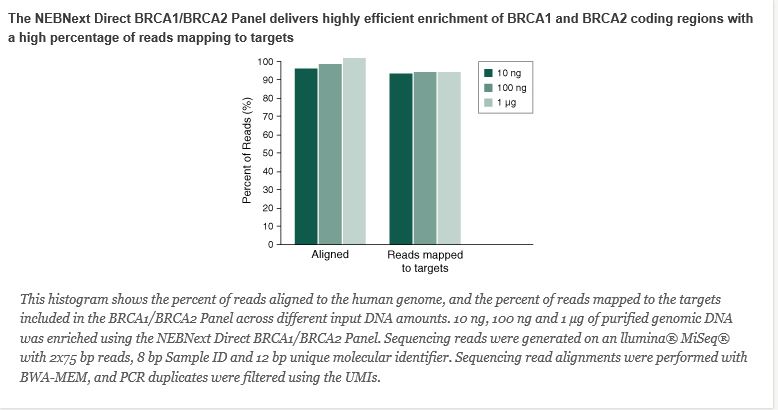
Generate full (100%) coverage of all protein coding regions in BRCA1 and BRCA2 genes
Obtain highly uniform sequencing across exon targets- 100% of base pairs in panel have coverage
greater than 20% of the mean target coverage
Maximize efficiency with a 1-day workflow that combines highly specific enrichment with library
preparation
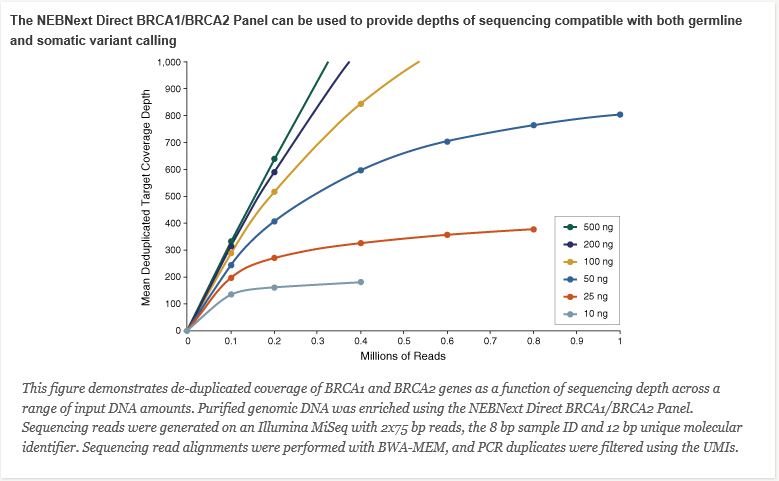
Produce high depths of target coverage across a wide range of DNA input amounts for germline and
somatic variant calling
The NEBNext Direct BRCA1/BRCA2 Panel enriches the complete exon content of BRCA1 and BRCA2
genes for next-generation sequencing analysis. NEBNext Direct employs a unique hybridization-based
enrichment workflow that hybridizes baits directly to genomic DNA, without the need for upfront library
preparation. The BRCA1/BRCA2 panel demonstrates extremely high specificity and unmatched coverage
uniformity across a wide range of DNA inputs, allowing highly sensitive calling of germline and somatic
variants while maximizing sequencer efficiency.
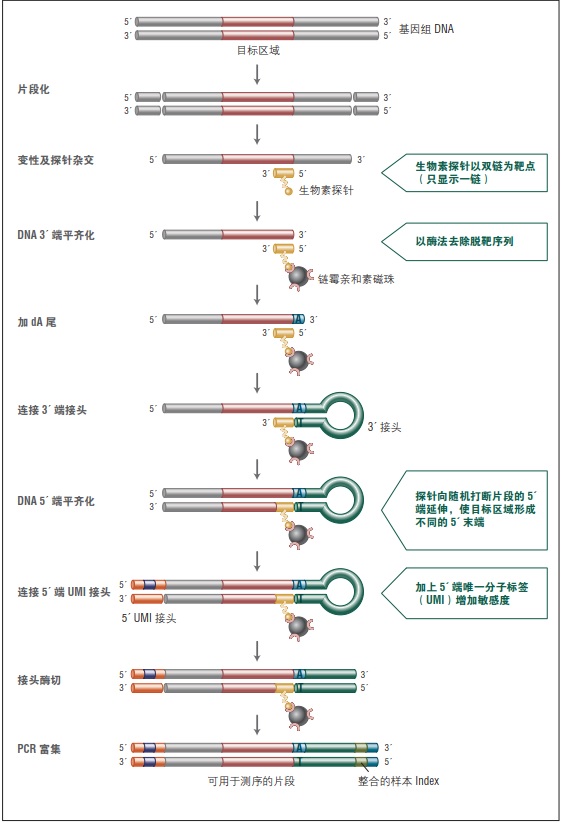
Figure 1. NEBNext Direct Workflow.
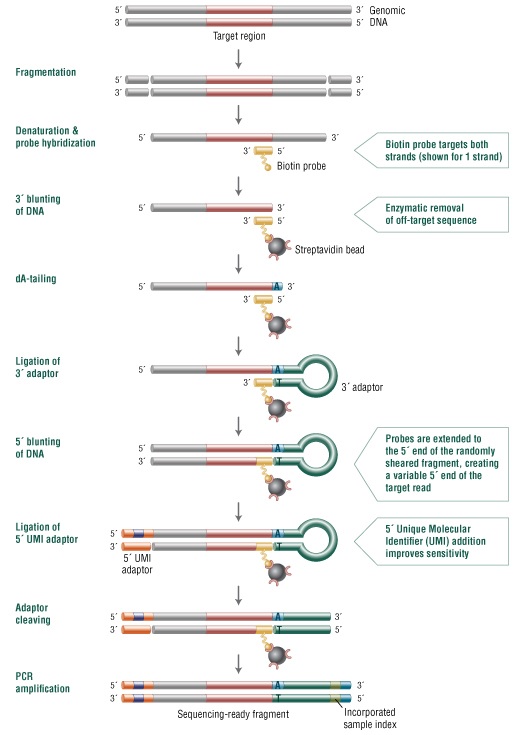
NEBNext Sample Purification Beads
NEBNext Direct BRCA1/BRCA2 Baits
NEBNext Direct® Bead Prep Buffer
NEBNext Direct® Hybridization Buffer
NEBNext Direct® dA-Tailing Buffer
NEBNext Direct® Adaptor Ligation Buffer
NEBNext Direct® Cleaving Buffer
NEBNext Direct® Hybridization Wash (HW)
NEBNext Direct® Bead Wash 1
NEBNext Direct® Bead Wash 2
NEBNext Direct® 3´ Adaptor
NEBNext Direct® 5´ UMI Adaptor
NEBNext Direct® dA-Tailing Enzyme
NEBNext Direct® Ligase
NEBNext Direct® 5´ Blunting Enzyme Mix
NEBNext Direct® Cleaving Enzyme Mix
NEBNext Direct® Q5 Master Mix
NEBNext Direct® FFPE Phosphorylation Enzyme
NEBNext Direct® Streptavidin Beads 4
NEBNext Direct® Hybridization Additive -20
NEBNext Direct® 5´ Blunting Buffer 4
NEBNext Direct® 3´ Blunting Enzyme Mix
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
可比对到目标区域的 reads 比例更高
所 有 区 域 均 可 得 到 均 一 的 测 序 结果,无论 GC 含量多少
将富集和文库制备流程相结合,1 天即可完成
能够从有限及降解的 DNA 样品中得到高质量的文库,包括 FFPE 及 ctDNA
区分分子重复,降低假阳性突变并提高敏感度
NEBNext Direct Cancer HotSpot Panel 采用了一种全新的靶向富集方法,使其能够高特异性的杂交捕获 50 个基因中的 190 个常见癌症靶点。NEBNext Direct 技术与传统的液相杂交及多重 PCR 方法相比有着明显的优势。靶向富集与文库制备相结合,减少了实验所需时间,同时将样本的损失降到最低。能够与自动化设备兼容的 NEBNext Direct 使得对难以成功的样本基因组中目标区域的深度测序成为可能,从而能够发现及鉴定样本中的罕见突变。
NEBNext Direct 采用了快速杂交实验流程,将捕获与文库构建相结合
目标区域包括以下癌症相关基因
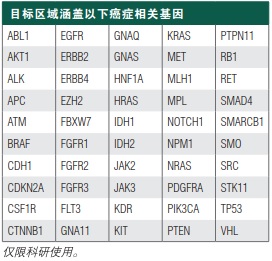
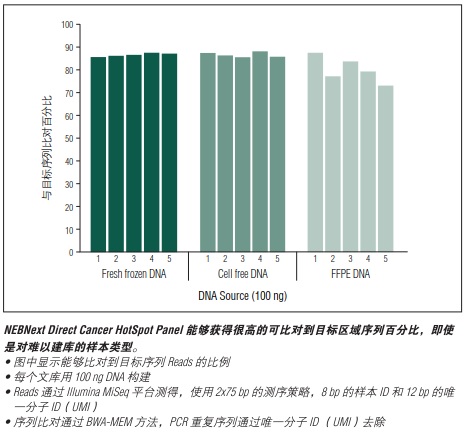

请登陆 NEBNextDirect.com 获取更多详细信息。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext Ultra II FS DNA 文库制备试剂盒 – 含片段化酶
NEBNext Ultra II FS DNA 文库制备试剂盒 – 含片段化酶/含纯化磁珠


除了每种组份都经过严格的质量监控以外,NEBNext DNA 试剂盒还都经过构建基因组 DNA 文库或 ChIP-Seq文库,再在 Illumina 测序平台测序进行验证。NEBNext 试剂盒里面,各个组份的批号也都被保留下来。注:接头、引物是单独供应的。

上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 酶学转化法甲基化建库相关产品
NEBNext 酶学转化法甲基化建库试剂盒
NEBNext 甲基化建库酶学转化法模块
NEBNext Q5U™ 预混液
NEBNext 酶学转化法甲基化建库多样本接头引物(双端检索引物)
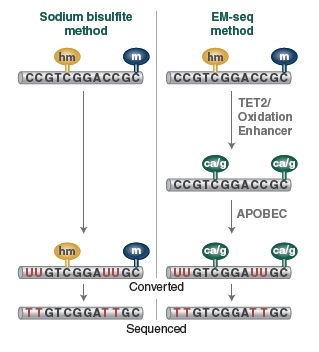
虽然重亚硫酸盐测序是研究 DNA 甲基化的金标准,但这种转化方式会对DNA 造成损伤,导致 DNA 断裂、丢失和 GC 偏嗜。NEBNext 酶学转化法甲基化建库试剂盒 (EM-seq™) 提供了一种酶学方法代替全基因组重亚硫酸盐处理 DNA (WGBS) 的方法,并结合高效流程化的建库步骤,适用于 Illumina 平台测序。高效的 EM-Seq 酶促转化可最大程度地减少对DNA的损伤,结合提供的 NEBNext Ultra II 文库制备流程,最终产生的高质量文库可以从有限的测序数据中更灵敏的检测到 5-mC 和 5-hmC。
更卓越的 5-mC 和 5-hmC 检测灵敏度
更高的比对率
更均一的 GC 覆盖度
能从有限的测序数据检测到更多的 CpG 位点
更均一的二核苷酸分布
更长的文库插入片段
更高效的建库流程
提供转化模块
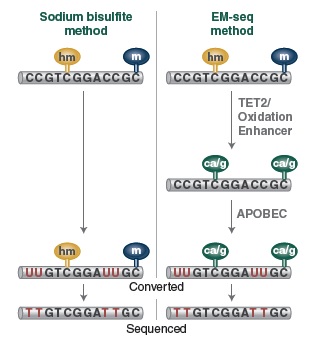
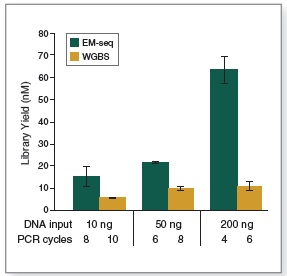
EM-Seq 能得到更高的文库产量。采用Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold 试剂盒进行重亚硫酸盐转化。上述所有起始量,EM-Seq 都能使用更少的 PCR 循环得到更高的文库产量,表明 EM-Seq 显著减少了 WGBS 方法中的 DNA 损失。误差条表示标准差。
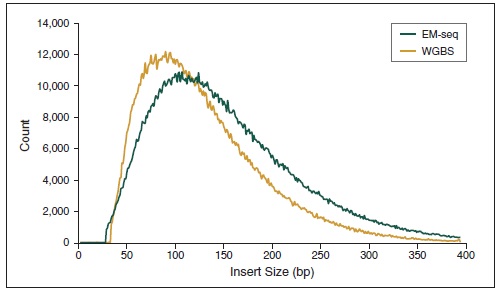
NEBNext 酶学转化法甲基化文库可获得更长的插入片段 。采用Covaris® S2 仪器将 50 ng 人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina MiSeq (2 X 76 bases) 测序,片段大小采用 Picard 2.18.14 测定。图中绘制了每个插入片段出现的频率标准化后的数据,结果如图:EM-Seq 建库后的插入片段比 WGBS 方法得到的插入片段更长,说明:酶学转化法不会对 DNA 造成损伤,而重亚硫酸盐处理的 DNA 有严重损伤。
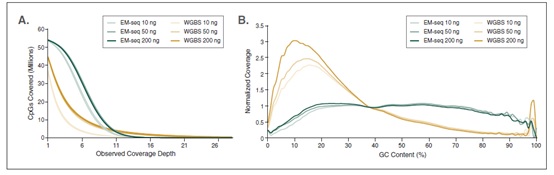
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点,并能实现更卓越的 GC 均一覆盖度。采用Covaris S2 仪器将 10 ng, 50 ng, 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000 (2 X 100 bases) 测序,使用 bwa-meth 0.2.2 将 测序数据 与 hg38 进行比对。
A: CpG 位点检测:通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度,其中每条链都独立计数,最终得到最多 5600 万个可能的 CpG 位点。结果显示:EM-Seq 在更低测序深度能检测到更多的 CpG 位点。
B: GC 覆盖度:使用 Picard 2.17.2 计算 GC 覆盖度,图中显示不同 GC 含量时 (0-100%),标准化后覆盖度的分布情况。结果显示:EM-Seq 文库显著提高 GC 覆盖度的均一性,无 AT 过度测序,也无 GC 测序不足,后两项都是 WGBS 文库的典型缺陷。
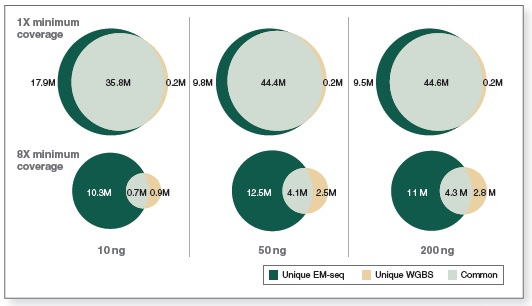
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点。采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000(2 X 100 bases)测序,使用 bwa-meth 0.2.2 将测序数据与 hg38 进行比对。通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度。
图中显示了 EM-Seq 和 WGBS 两种方法在不同起始量,至少 1X 和 8X 测序深度下检测到的独有和共有的 CpG 位点。EM-Seq 在至少 1X 测序深度下比 WGBS 多检测到 20% 以上的 CpG 位点。而在至少 8X 测序深度下,CpG 位点覆盖度差异增加至 2 倍。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 酶学转化法甲基化建库相关产品
NEBNext 酶学转化法甲基化建库试剂盒
NEBNext 甲基化建库酶学转化法模块
NEBNext Q5U™ 预混液
NEBNext 酶学转化法甲基化建库多样本接头引物(双端检索引物)
虽然重亚硫酸盐测序是研究 DNA 甲基化的金标准,但这种转化方式会对DNA 造成损伤,导致 DNA 断裂、丢失和 GC 偏嗜。NEBNext 酶学转化法甲基化建库试剂盒 (EM-seq™) 提供了一种酶学方法代替全基因组重亚硫酸盐处理 DNA (WGBS) 的方法,并结合高效流程化的建库步骤,适用于 Illumina 平台测序。高效的 EM-Seq 酶促转化可最大程度地减少对DNA的损伤,结合提供的 NEBNext Ultra II 文库制备流程,最终产生的高质量文库可以从有限的测序数据中更灵敏的检测到 5-mC 和 5-hmC。
更卓越的 5-mC 和 5-hmC 检测灵敏度
更高的比对率
更均一的 GC 覆盖度
能从有限的测序数据检测到更多的 CpG 位点
更均一的二核苷酸分布
更长的文库插入片段
更高效的建库流程
提供转化模块
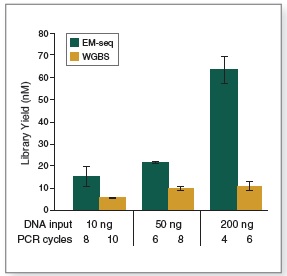
EM-Seq 能得到更高的文库产量。采用Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold 试剂盒进行重亚硫酸盐转化。上述所有起始量,EM-Seq 都能使用更少的 PCR 循环得到更高的文库产量,表明 EM-Seq 显著减少了 WGBS 方法中的 DNA 损失。误差条表示标准差。
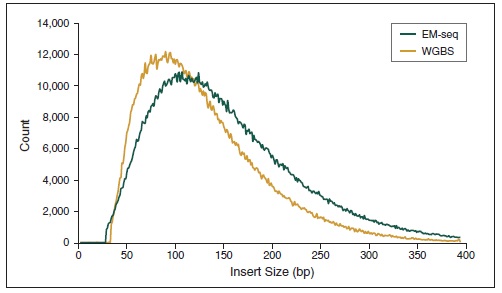
NEBNext 酶学转化法甲基化文库可获得更长的插入片段 。采用Covaris® S2 仪器将 50 ng 人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina MiSeq (2 X 76 bases) 测序,片段大小采用 Picard 2.18.14 测定。图中绘制了每个插入片段出现的频率标准化后的数据,结果如图:EM-Seq 建库后的插入片段比 WGBS 方法得到的插入片段更长,说明:酶学转化法不会对 DNA 造成损伤,而重亚硫酸盐处理的 DNA 有严重损伤。
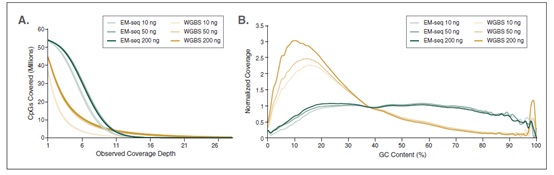
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点,并能实现更卓越的 GC 均一覆盖度。采用Covaris S2 仪器将 10 ng, 50 ng, 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000 (2 X 100 bases) 测序,使用 bwa-meth 0.2.2 将 测序数据 与 hg38 进行比对。
A: CpG 位点检测:通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度,其中每条链都独立计数,最终得到最多 5600 万个可能的 CpG 位点。结果显示:EM-Seq 在更低测序深度能检测到更多的 CpG 位点。
B: GC 覆盖度:使用 Picard 2.17.2 计算 GC 覆盖度,图中显示不同 GC 含量时 (0-100%),标准化后覆盖度的分布情况。结果显示:EM-Seq 文库显著提高 GC 覆盖度的均一性,无 AT 过度测序,也无 GC 测序不足,后两项都是 WGBS 文库的典型缺陷。
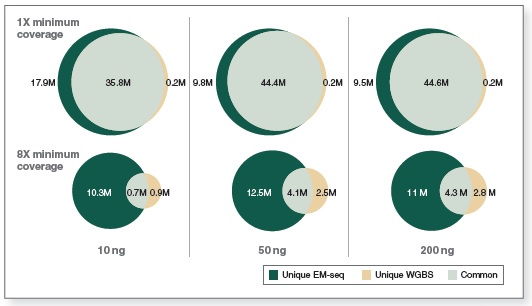
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点。采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000(2 X 100 bases)测序,使用 bwa-meth 0.2.2 将测序数据与 hg38 进行比对。通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度。
图中显示了 EM-Seq 和 WGBS 两种方法在不同起始量,至少 1X 和 8X 测序深度下检测到的独有和共有的 CpG 位点。EM-Seq 在至少 1X 测序深度下比 WGBS 多检测到 20% 以上的 CpG 位点。而在至少 8X 测序深度下,CpG 位点覆盖度差异增加至 2 倍。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 酶学转化法甲基化建库相关产品
NEBNext 酶学转化法甲基化建库试剂盒
NEBNext 甲基化建库酶学转化法模块
NEBNext Q5U™ 预混液
NEBNext 酶学转化法甲基化建库多样本接头引物(双端检索引物)
虽然重亚硫酸盐测序是研究 DNA 甲基化的金标准,但这种转化方式会对DNA 造成损伤,导致 DNA 断裂、丢失和 GC 偏嗜。NEBNext 酶学转化法甲基化建库试剂盒 (EM-seq™) 提供了一种酶学方法代替全基因组重亚硫酸盐处理 DNA (WGBS) 的方法,并结合高效流程化的建库步骤,适用于 Illumina 平台测序。高效的 EM-Seq 酶促转化可最大程度地减少对DNA的损伤,结合提供的 NEBNext Ultra II 文库制备流程,最终产生的高质量文库可以从有限的测序数据中更灵敏的检测到 5-mC 和 5-hmC。
更卓越的 5-mC 和 5-hmC 检测灵敏度
更高的比对率
更均一的 GC 覆盖度
能从有限的测序数据检测到更多的 CpG 位点
更均一的二核苷酸分布
更长的文库插入片段
更高效的建库流程
提供转化模块
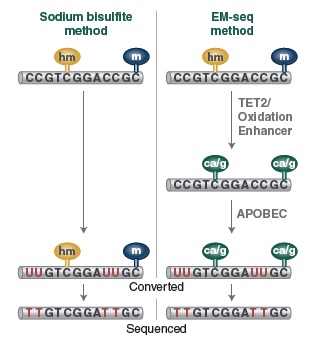
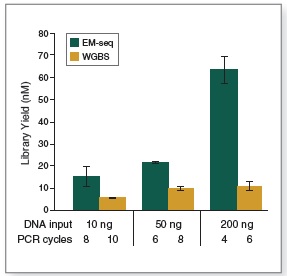
EM-Seq 能得到更高的文库产量。采用Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold 试剂盒进行重亚硫酸盐转化。上述所有起始量,EM-Seq 都能使用更少的 PCR 循环得到更高的文库产量,表明 EM-Seq 显著减少了 WGBS 方法中的 DNA 损失。误差条表示标准差。
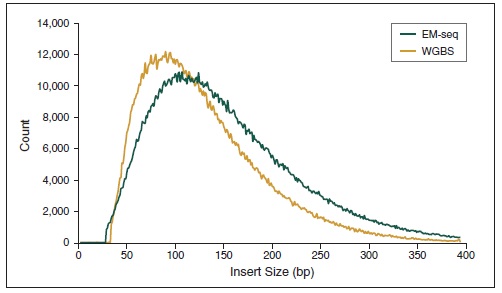
NEBNext 酶学转化法甲基化文库可获得更长的插入片段 。采用Covaris® S2 仪器将 50 ng 人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina MiSeq (2 X 76 bases) 测序,片段大小采用 Picard 2.18.14 测定。图中绘制了每个插入片段出现的频率标准化后的数据,结果如图:EM-Seq 建库后的插入片段比 WGBS 方法得到的插入片段更长,说明:酶学转化法不会对 DNA 造成损伤,而重亚硫酸盐处理的 DNA 有严重损伤。
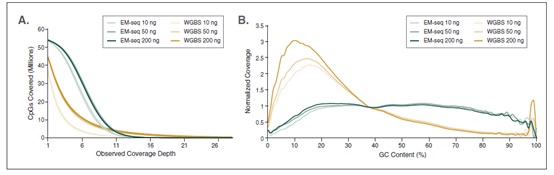
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点,并能实现更卓越的 GC 均一覆盖度。采用Covaris S2 仪器将 10 ng, 50 ng, 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000 (2 X 100 bases) 测序,使用 bwa-meth 0.2.2 将 测序数据 与 hg38 进行比对。
A: CpG 位点检测:通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度,其中每条链都独立计数,最终得到最多 5600 万个可能的 CpG 位点。结果显示:EM-Seq 在更低测序深度能检测到更多的 CpG 位点。
B: GC 覆盖度:使用 Picard 2.17.2 计算 GC 覆盖度,图中显示不同 GC 含量时 (0-100%),标准化后覆盖度的分布情况。结果显示:EM-Seq 文库显著提高 GC 覆盖度的均一性,无 AT 过度测序,也无 GC 测序不足,后两项都是 WGBS 文库的典型缺陷。
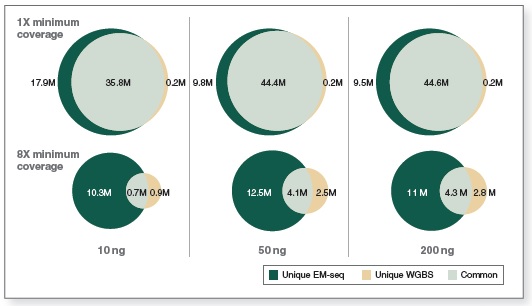
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点。采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000(2 X 100 bases)测序,使用 bwa-meth 0.2.2 将测序数据与 hg38 进行比对。通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度。
图中显示了 EM-Seq 和 WGBS 两种方法在不同起始量,至少 1X 和 8X 测序深度下检测到的独有和共有的 CpG 位点。EM-Seq 在至少 1X 测序深度下比 WGBS 多检测到 20% 以上的 CpG 位点。而在至少 8X 测序深度下,CpG 位点覆盖度差异增加至 2 倍。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext FFPE DNA 修复混合液
NEBNext Ultra II 末端修复/加 dA 尾模块
NEBNext 快速连接模块
NEBNext®-Oxford Nanopore Technologies® 连接测序配套试剂(E7180)包含以下三个 Oxford Nanopore Technologies 连接测序文库制备推荐模块:NEBNext FFPE DNA 修复混合液(M6630)、NEBNext Ultra II 末端修复/加 dA 尾模块(E7546)和 NEBNext 快速连接模块(E6056)。现在您可更方便地通过同一个货号 E7180 订购以上所有模块,并且该产品已为 Oxford Nanopore Technologies 连接测序试剂盒(SQK-LSK109)优化了体积。
为连接测序试剂盒(SQK-LSK109)进行了组份体积的优化
更简化的订购和库存管理
适用于所有 Nanopore 测序仪:Minion®、Gridion®、Promethion®、Floungle®
杜绝浪费——没有多余的缓冲液或过量的试剂
NEBNext® FFPE DNA 修复混合液(0.048 ml)
NEBNext® FFPE DNA 修复缓冲液(0.084 ml)
NEBNext® Ultra™ II 末端修复酶混合液(0.072 ml)
NEBNext® Ultra™ II 末端修复反应缓冲液(0.084 ml)
快速 T4 连接酶(0.024 ml)
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
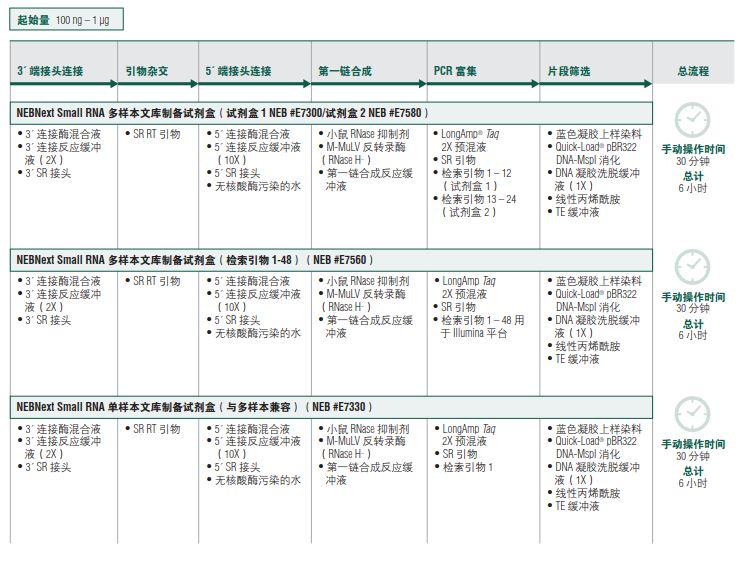
NEBNext Small RNA 多样本文库制备试剂盒 2
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext Small RNA 多样本文库制备试剂盒 1
NEBNext Small RNA 多样本文库制备试剂盒 2
ssDNA 接头腺苷酸化的客户,请选用 5´ DNA 腺苷化试剂盒
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
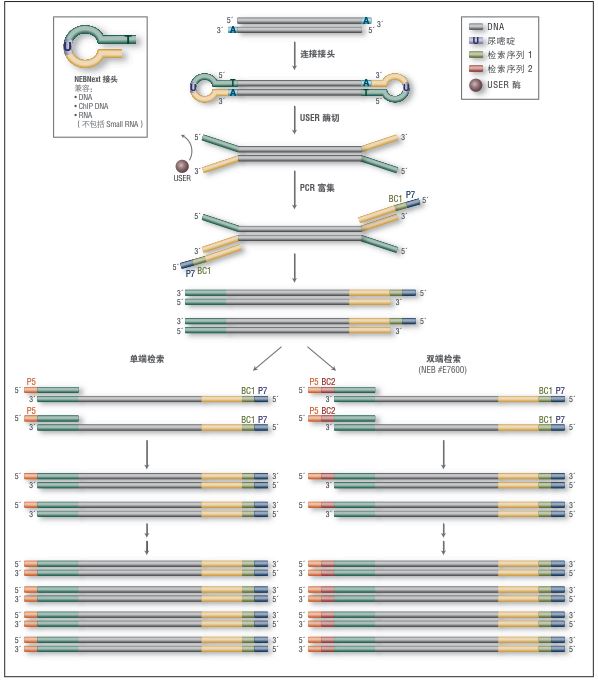
每个试剂盒都通过对基因组 DNA、ChIP DNA 和 mRNA 进行文库构建,并在 Illumina 平台测序进行验证。
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
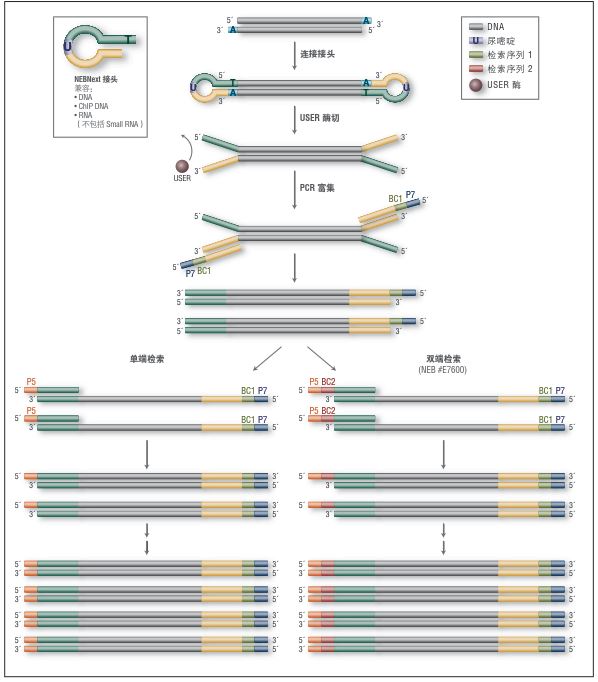
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
该新产品进一步提升了二代测序(NGS)流程中 FFPE DNA 的修复效果
FFPE 是福尔马林固定石蜡包埋样本,由于其固定和保存的方法,给其中的 DNA 造成严重损伤,因此从 FFPE 样本中获取高质量的序列数据就变得具有挑战性。NEBNext® FFPE DNA 修复模块 v2 是一种优化的酶混合物,旨在修复 FFPE 样本 DNA,同时配有经过优化的试剂,能实现更精简的 NGS 文库制备流程。
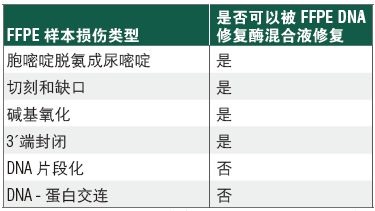

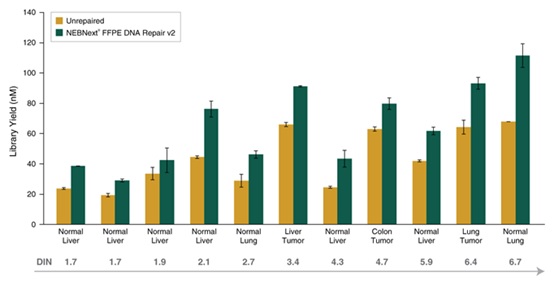
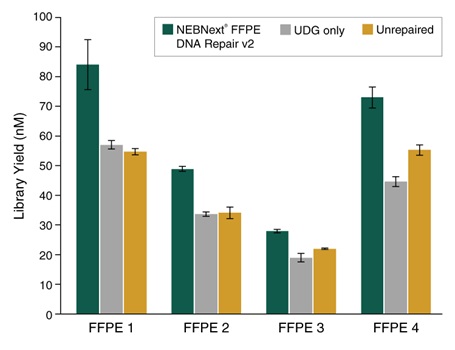
NEBNext® FFPE DNA 修复模块 v2 修复不同质量的 FFPE DNA 后,都能进行高效的文库制备。选用 25 ng 不同质量和组织来源的 Covaris® 超声打断后的 FFPE DNA 样本制备文库。使用 NEBNext® FFPE DNA 修复模块 v2 修复后使用 NEBNext® Ultra™ II DNA 文库制备试剂盒(NEB #E7645)建库,共进行 9 个 PCR 循环。使用 Agilent® HS D1000 TapeStation® 进行文库定量。NEBNext® FFPE DNA 修复模块 v2 根据起始 DNA 的质量和损伤类型,不同程度地提高 FFPE 文库的产量。误差条表示每个文库样本两次重复的标准差。
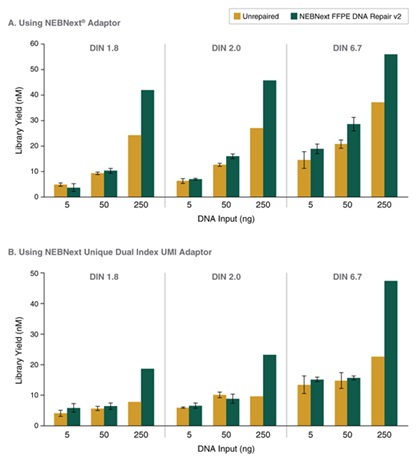
使用 NEBNext® 发夹结构接头和 Unique 双端 Index 引物,UMI 接头,NEBNext® FFPE DNA 修复模块 v2 修复 5 – 250 ng FFPE DNA 后,都能进行高效的文库制备。选用 5 ng、50 ng 和 250 ng 三种不同质量的正常肝脏 FFPE DNA 样本制备文库,比较使用和不使用 NEBNext® FFPE DNA 修复模块 v2 修复的文库制备效果。使用 NEBNext® Ultra™ II DNA 文库制备试剂盒(NEB #E7645)和(a)NEBNext® 发夹结构接头,针对 5 ng、50 ng 和 250 ng 三种起始 DNA 分别使用 11、8 和 6 个 PCR 循环制备文库,以及(b)NEBNext® Unique 双端 Index 引物,UMI 接头(NEB #E7395),针对 5 ng、50 ng 和 250 ng 三种起始 DNA 分别使用 11、8 和 6 个 PCR 循环制备文库。使用 Agilent® HS D1000 TapeStation® 进行文库定量,图中绘制了 2 个文库(5 和 50 ng)和 1 个重复(250 ng)的平均产量。误差条表示 5 和 50 ng 文库重复的标准差。使用 NEBNext® 发夹结构接头和 Unique 双端 Index 引物,UMI 接头,针对 5 ng 至 250 ng 的 FFPE DNA 样本,NEBNext® FFPE DNA 修复模块 v2 都能进行高效的文库制备。
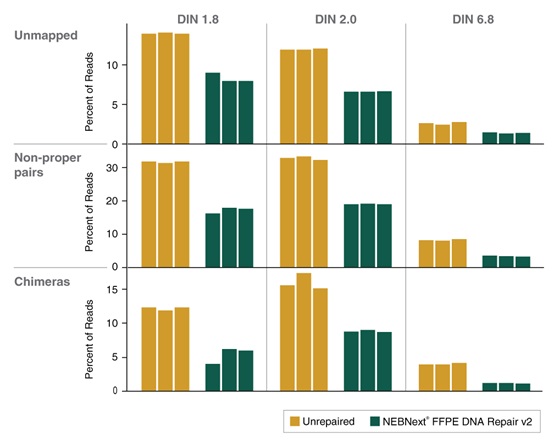
NEBNext® FFPE DNA 修复模块 v2 能提高各项文库质量指标,包括比对率、正确匹配的双端 reads 和嵌合序列。选用三份 50 ng 不同质量的正常肝脏 FFPE DNA 样本(使用或未使用 NEBNext® FFPE DNA 修复模块 v2 修复),采用 NEBNext® Ultra™ II DNA 文库制备试剂盒(NEB #E7645)制备文库。使用 Illumina NextSeq® 500 测序。向下抽样 100 万个双端 Reads,并使用 Bowtie2(v2.3.2)将 Reads 与 GRCh38 人类参考基因组(RefSeq 884148)进行比对。使用 MarkDuplicates(v1.56.0)分析比对上的 Reads,使用 Picard SAM/BAM 比对汇总指标(v1.56.0)。使用 NEBNext® FFPE DNA 修复模块 v2 可提高比对率,并降低非正确匹配和嵌合序列。
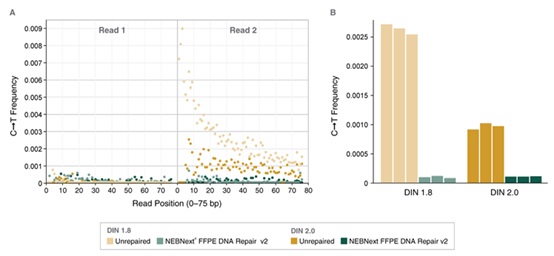
NEBNext® FFPE DNA 修复模块 v2 能修复 FFPE DNA 样本中存在的大量胞嘧啶脱氨损伤。选用两种 50 ng 不同质量的正常肝脏 FFPE DNA 样本(DIN 2.0 和 DIN 1.8,),使用或未使用 NEBNext® FFPE DNA 修复模块 v2 修复后,采用 NEBNext® Ultra™ II DNA 文库制备试剂盒(NEB #E7645)制备文库后,使用 Illumina NextSeq® 500 测序。向下抽样 100 万个双端 Reads,并使用 Bowtie2(v2.3.2)将 Reads 与 GRCh38 人类参考基因组(RefSeq 884148)进行比对。根据等式MAX([C T]-[G A])/([NC]+[NG]),EXP(-10)),使用 MarkDuplicates(v1.56.0)分析比对上的 Reads,使用 Tasmanian(V0.1.3)分析 dCdT(CT)突变。(a):在 Read 1 和 Read 2 中,绘制了 CT 突变频率与 Read 位置(0 – 75 bp)的函数关系图。当使用 NEBNext® FFPE DNA 修复模块 v2 修复后,在两个不同的 FFPE DNA 样本中,这些 CT 突变的丰度和位置偏差都显著降低。(b):Read 2 中的错误突变被量化为总频率。图中结果是每种情况下的两个重复数据。

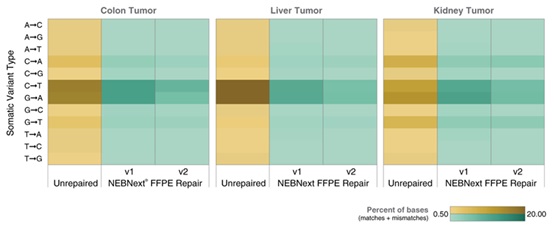
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
停产通知:该产品将于 2021 年 12 月 15 日停产,推荐替代产品为 #E7645S
NEBNext DNA Ultra 文库制备试剂盒适用于 Illumina 测序平台。建议样品起始量总 DNA 为 5 ng – 1 µg,使用优化的建库流程。请注意,接头和引物不包含在试剂盒内,需单独购买。
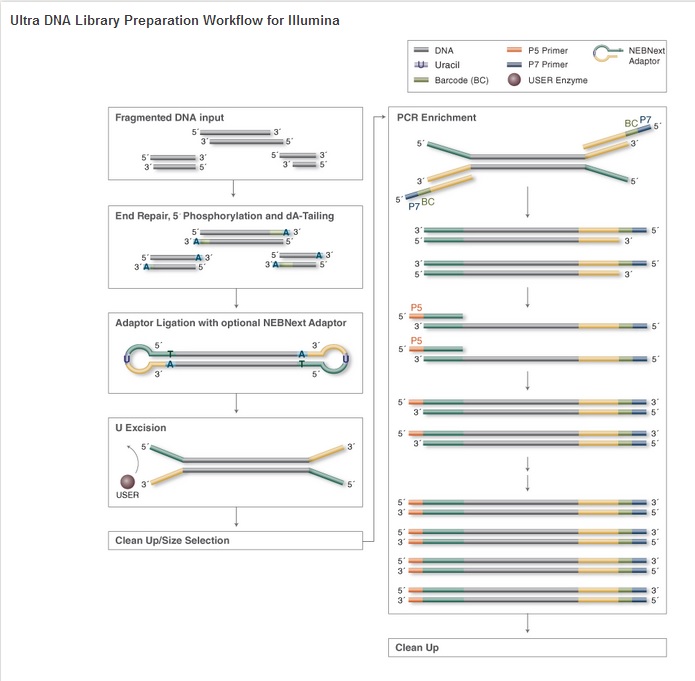
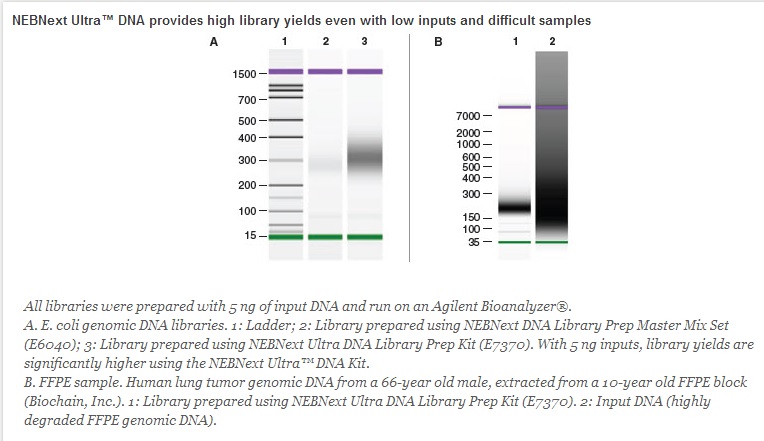
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。